组学测序

定义:
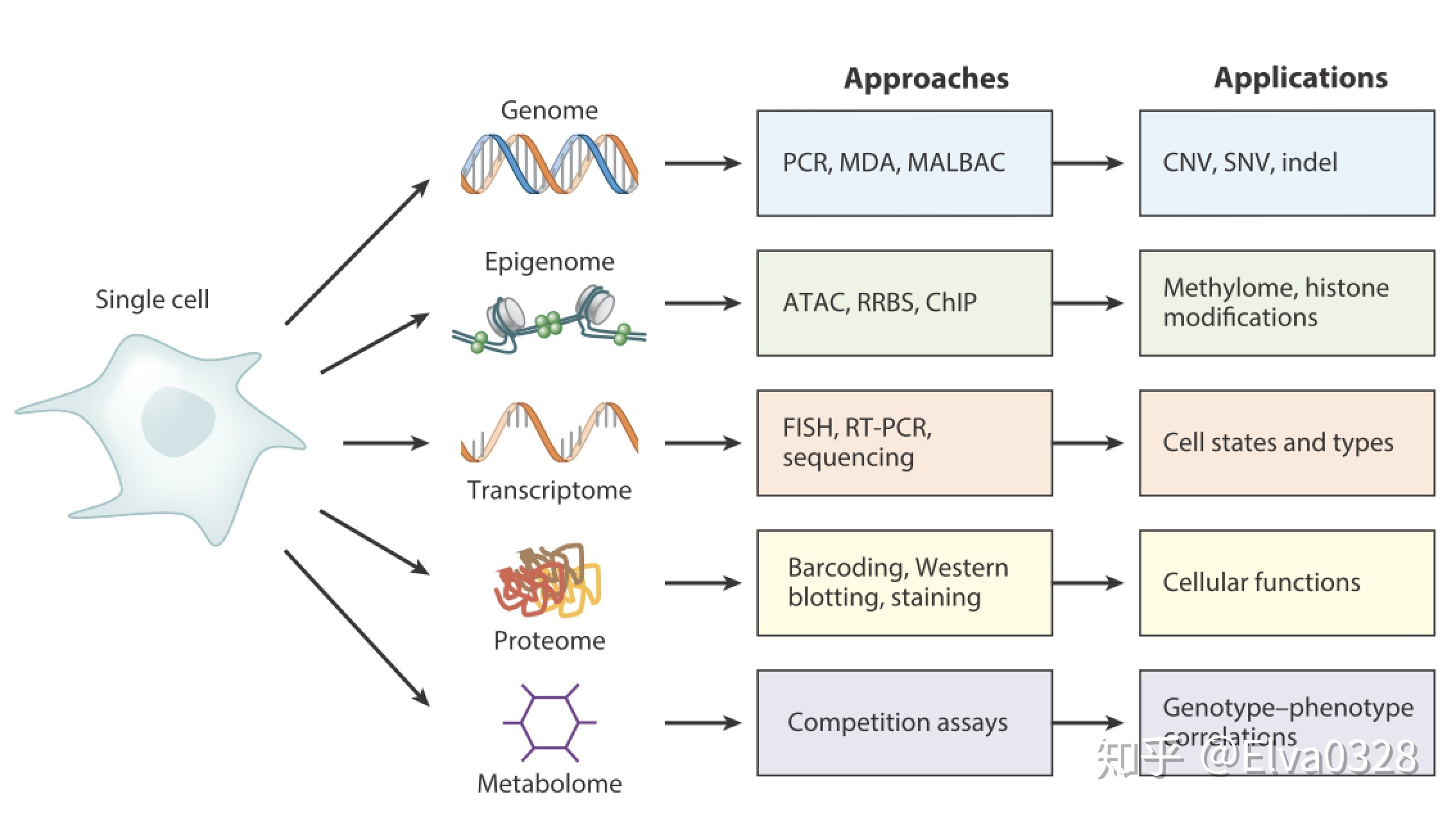
是指在单个细胞水平上对转录组或基因组进行扩增并测序,以检测单细胞在基因组(结构变异-Structural Variations-SVs;拷贝数变异-Copy number variants-CNVs;单核苷酸变异-Single nucleotide variants-SNVs等),转录组学(RNA表达水平;转录本的选择性剪接),表观组学(DNA 甲基化等),蛋白组学多个组学的数据。

1. DNA的序列:ATCG怎么排列,以及各序列的丰度;
2. DNA的表观遗传修饰:比如甲基化、羟甲基化,以及组蛋白的各种修饰;
3. RNA的序列:AUCG怎么排列,以及各序列的丰度;
4. RNA的表观遗传修饰:比如近年很火的m6A修饰;
5. 染色质的结构:3C、4C、5C等各种C;
6. 其他应用:比如DNA损伤位置、蛋白-蛋白相互作用等。
单细胞表达谱测序技术可以从混杂组织中捕获单个细胞,并能从中够获取单个细胞的表达信息。
2. WHY 为什么要使用单细胞测序
遗传信息的异质性:
单个细胞是生命活动的最终单元,其中遗传机制和细胞环境相互作用并形成组织和器官等复杂结构的形成和功能。传统研究方法是在多细胞水平进行的,因此最终得到的信号值,其实是多个细胞的平均,丢失了异质性的信息。
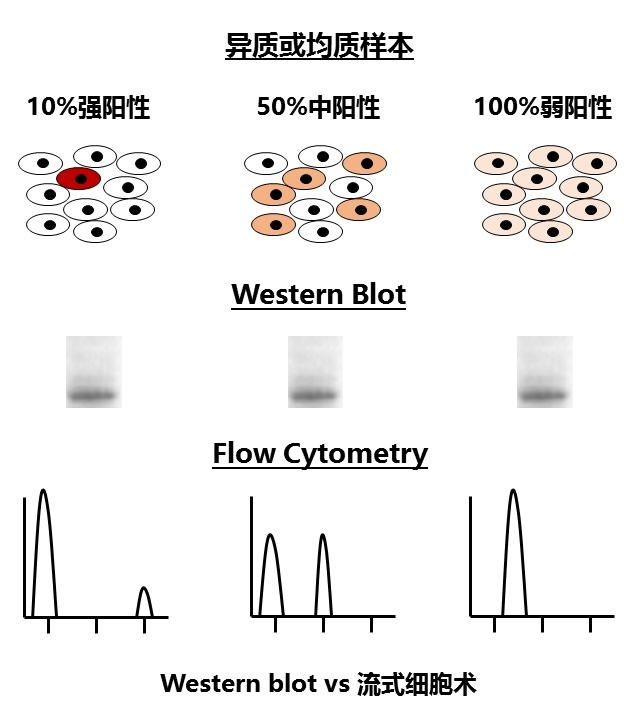
为检测某个蛋白质的表达量,可以用Western blot和流式细胞术来实现。但是,WB的话,并没有办法区分以下情况:目的蛋白只在10%的细胞中强表达,还是在50%的细胞里中等表达,还是在所有细胞中弱表达呢?因为最终电泳跑出来,就是一条差不多强度的带——但如果用流式细胞术这种在单细胞水平对荧光强度加以测定的技术,就能区分上述的情况了。同样道理,单细胞测序能够检出混杂样品测序所无法得到的异质性信息。
3. HOW 如何实现单细胞测序
3. HOW 如何实现单细胞测序
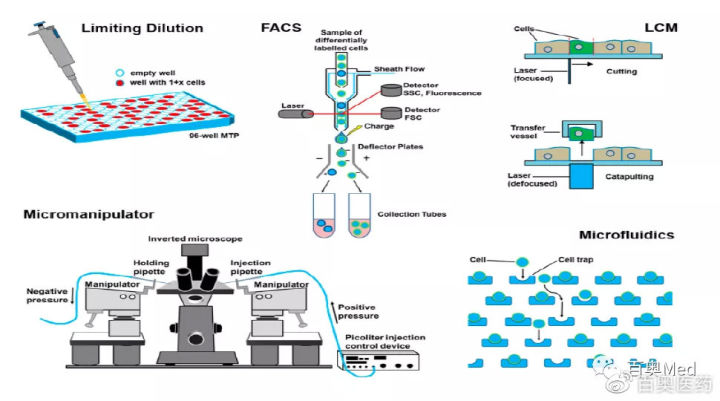
早期的单细胞获取方式,是通过一定的技术手段,将单个细胞分离出来,并利用SMART-Seq2或商业化的SMART-Seq4试剂独立构建测序文库,最终进行测序。捕获方式包括:有限稀释法,流式分选法,激光切割法,显微操作法(图中前四种)。
这些方法虽然各有各的优点,但各自的缺点也十分明显,比如流式分选法对于难度的计算易存在误差;流式分选法不适用于微量样本;激光切割法操作复杂;显微切割法的细胞通量低等。
通过微流控芯片将单个细胞捕获至油滴(图中最后一种),就是微流控技术。这种方式的优点是细胞通量高,周期快速,成本低,细胞捕获效率高,并且商业化仪器操作简便。其核心思想是将不同的细胞赋予一段不同的barcode序列,建库时,携带相同barcode序列的核酸分子被认为来自同一个细胞,人们就可以一次性为成百上千的细胞建库并顺利区分它们。
这些方法虽然各有各的优点,但各自的缺点也十分明显,比如流式分选法对于难度的计算易存在误差;流式分选法不适用于微量样本;激光切割法操作复杂;显微切割法的细胞通量低等。
通过微流控芯片将单个细胞捕获至油滴(图中最后一种),就是微流控技术。这种方式的优点是细胞通量高,周期快速,成本低,细胞捕获效率高,并且商业化仪器操作简便。其核心思想是将不同的细胞赋予一段不同的barcode序列,建库时,携带相同barcode序列的核酸分子被认为来自同一个细胞,人们就可以一次性为成百上千的细胞建库并顺利区分它们。
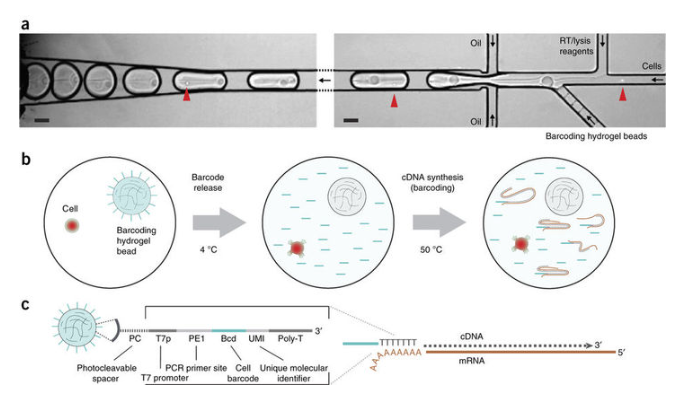
当测序对象是mRNA:
由于mRNA测序前需要做逆转录,只需要在poly T引物的5’端加入barcode即可。首先将单细胞悬液样品和带有barcode的水凝胶珠子,通过微流体芯片,包裹在一个油滴之中。在油滴中进行逆转录之后,每一个单细胞的cDNA文库,就带上了独一无二的barcode(蓝色部分)。最后再将所有的单细胞cDNA文库混在一起测序,通过程序识别barcode,区分单细胞。
由于mRNA测序前需要做逆转录,只需要在poly T引物的5’端加入barcode即可。首先将单细胞悬液样品和带有barcode的水凝胶珠子,通过微流体芯片,包裹在一个油滴之中。在油滴中进行逆转录之后,每一个单细胞的cDNA文库,就带上了独一无二的barcode(蓝色部分)。最后再将所有的单细胞cDNA文库混在一起测序,通过程序识别barcode,区分单细胞。
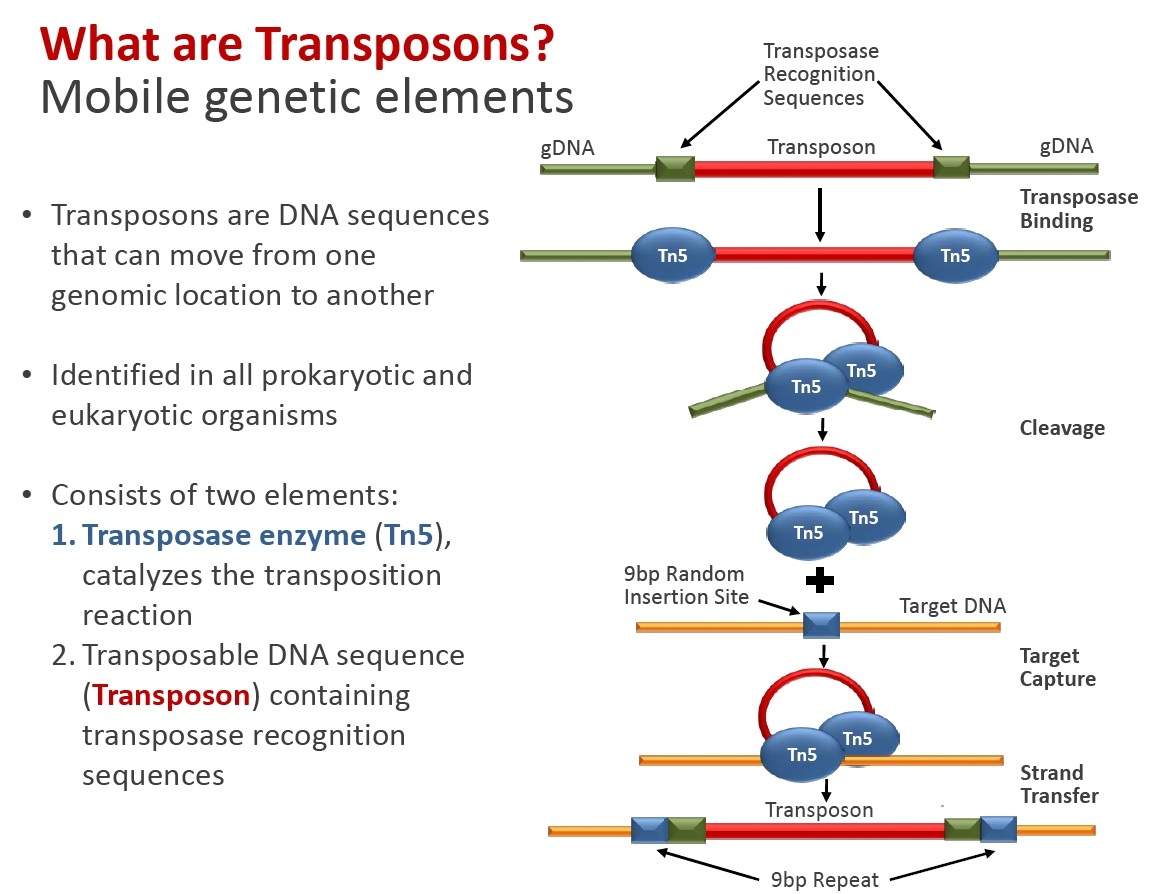
当测序对象是DNA:
如果测序对象是DNA,比如全基因组,就需要用别的方式来加barcode。目前主要是通过一种经过改造的高效转座酶(transposase)Tn5来实现。基因转座是指转座子DNA从一个染色体座位“跳跃”到另外一个座位的过程。单细胞的DNA测序就利用了这个特性,将barcode DNA预先和转座酶Tn5组装好,再通过上述的微流体技术,将细胞和转座复合物包裹在一个油滴之中。随后,转座酶会把barcode插入到基因组DNA之中。这个过程在文献中也被成为tagmentation。
具体操作过程中为提高效率,通常走组合索引(combinatorial indexing)路线:将单细胞悬液放在多孔板中,并用转座酶Tn5给细胞加第一个barcode,这里每个孔中的barcode是不同的。然后,再将样品混合起来,通过流式细胞术,将少量的细胞分选到含有建库PCR引物的多孔板中。而这些引物是带有第二轮barcode的。因此,经过Tn5的转座,和PCR加标签,绝大部分的细胞就能带上独一无二的barcode了。
目前,大规模被使用的单细胞测序平台主要有以下五种:
1. 10x Chromium Single Cell Gene Expression Solution;
2. BD Rhapsody™ Single-Cell Analysis System;
3. Illumina® Bio-Rad® Single-Cell Sequencing Solution;
4. ICELL8 Single-Cell System;
5.C1™ 单细胞全自动制备系统。
其中前两种为主流。
如果测序对象是DNA,比如全基因组,就需要用别的方式来加barcode。目前主要是通过一种经过改造的高效转座酶(transposase)Tn5来实现。基因转座是指转座子DNA从一个染色体座位“跳跃”到另外一个座位的过程。单细胞的DNA测序就利用了这个特性,将barcode DNA预先和转座酶Tn5组装好,再通过上述的微流体技术,将细胞和转座复合物包裹在一个油滴之中。随后,转座酶会把barcode插入到基因组DNA之中。这个过程在文献中也被成为tagmentation。
具体操作过程中为提高效率,通常走组合索引(combinatorial indexing)路线:将单细胞悬液放在多孔板中,并用转座酶Tn5给细胞加第一个barcode,这里每个孔中的barcode是不同的。然后,再将样品混合起来,通过流式细胞术,将少量的细胞分选到含有建库PCR引物的多孔板中。而这些引物是带有第二轮barcode的。因此,经过Tn5的转座,和PCR加标签,绝大部分的细胞就能带上独一无二的barcode了。
目前,大规模被使用的单细胞测序平台主要有以下五种:
1. 10x Chromium Single Cell Gene Expression Solution;
2. BD Rhapsody™ Single-Cell Analysis System;
3. Illumina® Bio-Rad® Single-Cell Sequencing Solution;
4. ICELL8 Single-Cell System;
5.C1™ 单细胞全自动制备系统。
其中前两种为主流。

10x Chromium Single Cell Gene Expression Solution
可通过快速高效的单细胞标记、测序和分析,获得单细胞水平的基因表达谱,对复杂细胞群体进行深入细致分析,绘制大规模单细胞表达图谱。通量高,周期快速,成本低,细胞捕获效率高,并且商业化仪器操作简便。但其对细胞活性要求较高,大于90%。
缺点:
对细胞总量及活性要求较高,需仪器上门服务;
3’测序,基因检测率不及SMART-seq等全长测序;缺乏质控点;平台环境较为封闭,个性化研发成本较高。
适用范围:
直径40μm以下细胞(受仪器管道直径限制);
可通过快速高效的单细胞标记、测序和分析,获得单细胞水平的基因表达谱,对复杂细胞群体进行深入细致分析,绘制大规模单细胞表达图谱。通量高,周期快速,成本低,细胞捕获效率高,并且商业化仪器操作简便。但其对细胞活性要求较高,大于90%。
缺点:
对细胞总量及活性要求较高,需仪器上门服务;
3’测序,基因检测率不及SMART-seq等全长测序;缺乏质控点;平台环境较为封闭,个性化研发成本较高。
适用范围:
直径40μm以下细胞(受仪器管道直径限制);
大规模细胞样本,适用于分群、谱系等研究。
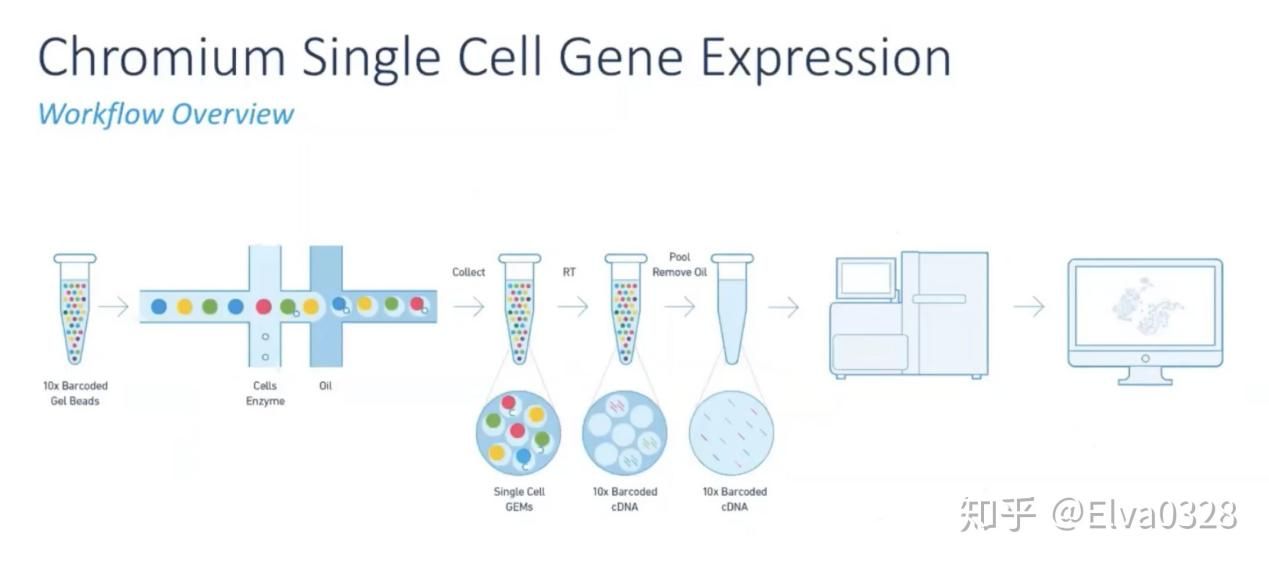

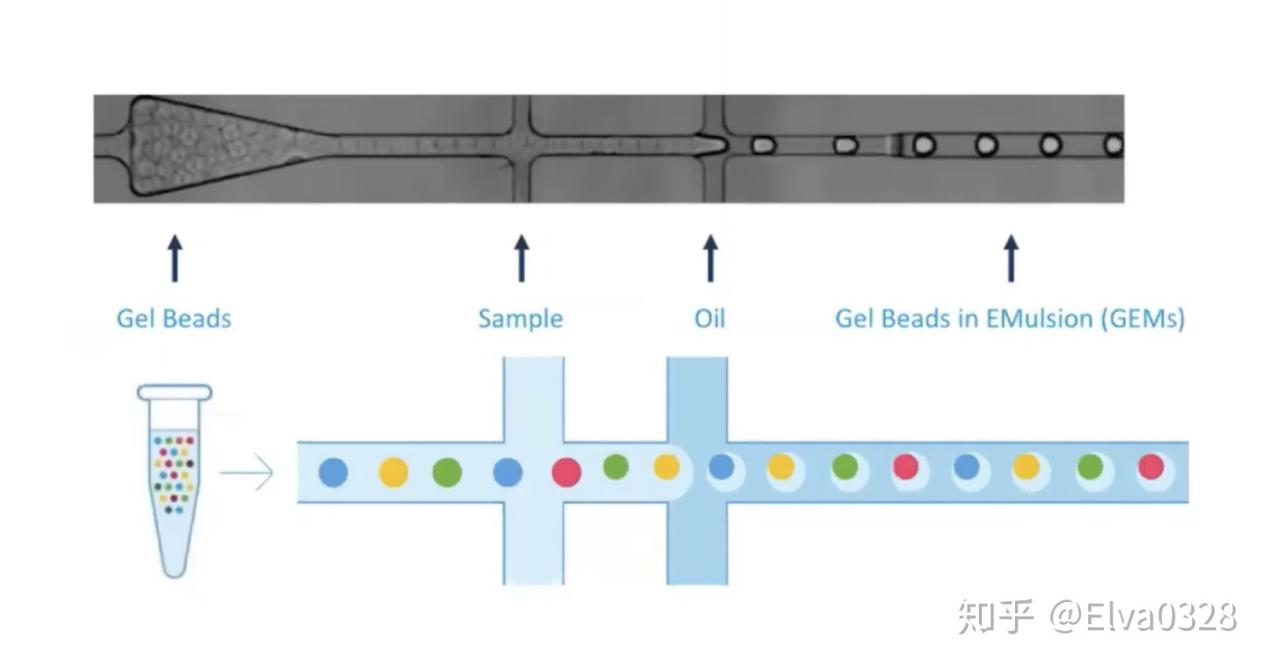
工作原理:
1. 将单个细胞与含有条形码(barcode)和引物(umi+ploy(dT)VN)的凝胶珠包裹在一个油滴。从横向孔道中逐一输入凝胶微珠,第一纵向孔道输入细胞,凝胶微珠与细胞碰撞后会吸附在凝胶微珠上,并通过微流控技术,将之输入到第二纵向孔道,即油相孔道中。这时候,就形成了一个个油滴,最终输出并收集在EP管中。每一个油滴中会落入一个细胞以及一个凝胶微珠,那么在每一个凝胶微珠中上长满了不同的Cell Barcode和UMI Barcode连接形成的序列,再加上一端PolyT的抓手,构成我们的捕获凝胶微珠。而这个凝胶微珠抓手就会使用oligo dT抓住mRNA构建文库。该过程可使细胞标记上特殊的barcode,从而达到单细胞测序的目的。
2. 之后在每个油滴内,凝胶珠溶解,细胞裂解释放mRNA,通过逆转录产生用于测序的带条形码的cDNA;
液体油层破坏后,cDNA后续进行文库构建,使用Illumina测序平台对文库进行测序检测,即可一次性获得大量单细胞的基因表达数据,10min内自动完成多至80,000个细胞的捕获,从而达到在单细胞水平进行表达测序的目的。
1. 将单个细胞与含有条形码(barcode)和引物(umi+ploy(dT)VN)的凝胶珠包裹在一个油滴。从横向孔道中逐一输入凝胶微珠,第一纵向孔道输入细胞,凝胶微珠与细胞碰撞后会吸附在凝胶微珠上,并通过微流控技术,将之输入到第二纵向孔道,即油相孔道中。这时候,就形成了一个个油滴,最终输出并收集在EP管中。每一个油滴中会落入一个细胞以及一个凝胶微珠,那么在每一个凝胶微珠中上长满了不同的Cell Barcode和UMI Barcode连接形成的序列,再加上一端PolyT的抓手,构成我们的捕获凝胶微珠。而这个凝胶微珠抓手就会使用oligo dT抓住mRNA构建文库。该过程可使细胞标记上特殊的barcode,从而达到单细胞测序的目的。
2. 之后在每个油滴内,凝胶珠溶解,细胞裂解释放mRNA,通过逆转录产生用于测序的带条形码的cDNA;
液体油层破坏后,cDNA后续进行文库构建,使用Illumina测序平台对文库进行测序检测,即可一次性获得大量单细胞的基因表达数据,10min内自动完成多至80,000个细胞的捕获,从而达到在单细胞水平进行表达测序的目的。

BD Rhapsody™ Single-Cell Analysis System
Cytoseq分子标签技术,能为单细胞中每个转录本标记特异性分子标签,实现单细胞水平上基因表达谱的绝对定量。每个细胞也会被标记特异性细胞标签,结合Rhapsody特有的单细胞分离技术,单次实验可制备100-10000个单细胞文库,可根据需求定制引物。
缺点:
对细胞总量要求较高;为3’测序,基因检测率不及SMART-seq等全长测序;操作步骤较多。
适用范围:
直径<20μm的细胞(受磁珠结合效率限制)。
大规模细胞样本,适用于分群、谱系等研究。
Cytoseq分子标签技术,能为单细胞中每个转录本标记特异性分子标签,实现单细胞水平上基因表达谱的绝对定量。每个细胞也会被标记特异性细胞标签,结合Rhapsody特有的单细胞分离技术,单次实验可制备100-10000个单细胞文库,可根据需求定制引物。
缺点:
对细胞总量要求较高;为3’测序,基因检测率不及SMART-seq等全长测序;操作步骤较多。
适用范围:
直径<20μm的细胞(受磁珠结合效率限制)。
大规模细胞样本,适用于分群、谱系等研究。
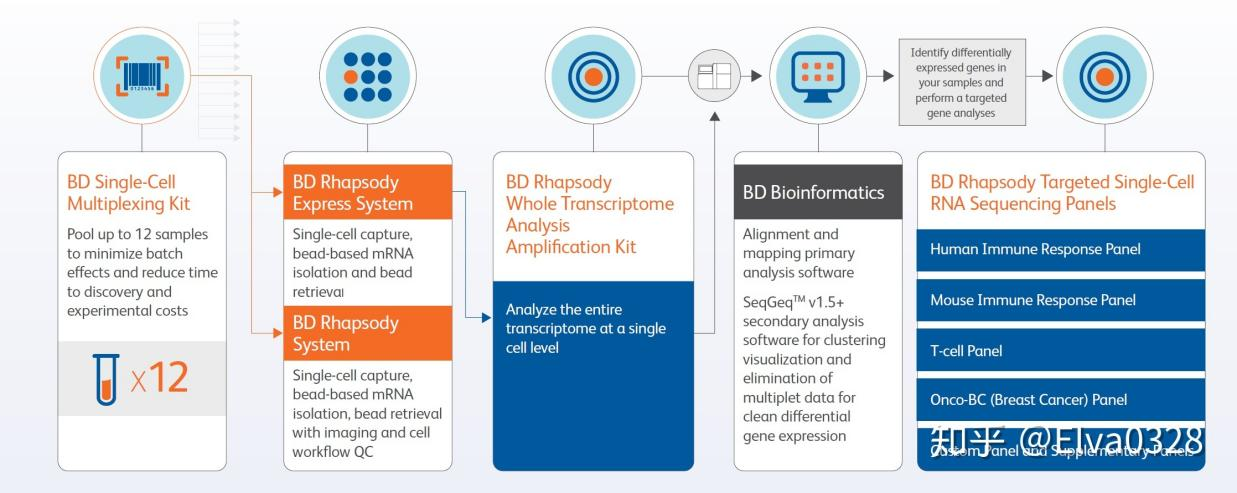
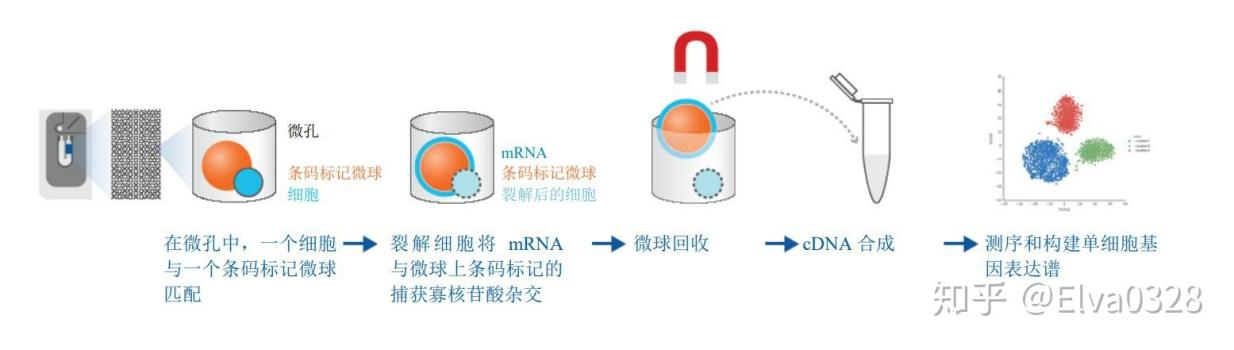
工作原理:
BD Rhapsody™ Single-Cell Analysis System的扩增和测序部分和10 x Chromium Single Cell Gene Expression Solution是一样的,两者的区别主要在于捕获单细胞的部分。
如图所示,这是BD捕获单细胞的方法。首先它使用的不是基于微流控芯片技术的双十字系统,而是蜂巢板。蜂巢板上每个孔的大小比一个细胞稍微大一些,可以同时放下一个细胞和一个凝胶微珠。它的操作是细胞悬液经注入孔注入后,自然沉降到反应孔中,随后, Beads 同样由注入孔注入,即可在单个反应孔中捕获其中的细胞。这里的 Beads 是刚性 Beads,捕获后在低温条件下可以保存3个月。Beads 上的序列结构与 10X Genomics 相似,可以捕获到游离 mRNA 的 polyA 尾。然后洗去多余的凝胶微珠,从而达到单细胞捕获的目的。
但是这样的操作会导致有的蜂巢孔是空的,而有的进去两个细胞,所以该系统装备了成像系统,在细胞捕获后,即可直观的看到细胞捕获数、细胞状态等信息。当这些统计的指标都是PASS的时候,就说明此次单细胞捕获是合格的,可以进行后续的操作了。当细胞捕获后,它的后续部分原理和10X就一样了。
数据处理:
标准预处理
1、QC和选择细胞进行进一步分析
2、规范化数据
3、识别高度可变的特征(特征选择)
4、缩放数据
主成分分析(PCA)
1、线性降维
2、确定数据集的“维度”
聚类细胞
数据呈现:
非线性降维(UMAP/tSNE)对低维度数据可视化处理,反映原始数据结构
寻找差异表达的特征(簇生物标志物),可视化比较某个基因在几个cluster之间的表达量
1. APPLICATION 单细胞测序的应用
单细胞转录组测序(scRNA-seq)
单细胞测序(scRNA-seq)对于制备的细胞悬液的细胞活性和细胞数目有着较高的要求,而且必须要求新鲜的样本,但是那些已经冻起来的样本库里面大量的珍贵样本,比如脑组织,心脏组织,肿瘤组织等都无法进行scRNA-seq,单细胞核转录组测序的出现极大的解决了这个问题。
单细胞核转录组测序(snRNA-seq)
单细胞核转录组测序(Single Nuclei RNA Sequencing,snRNA-seq):通过提取样本单细胞核,然后分离、标记细胞核,在单细胞水平研究核基因表达检测的技术。为脑组织、心脏、肾脏等复杂组织或一些珍稀冻存样品提供了单细胞水平研究应用平台,可挖掘更多潜在的致病细胞类型,更易于探讨肿瘤细胞异质性和致病机理。对免疫细胞类型不友好。
单细胞免疫组库测序
10X Genomics Single Cell Immune Profiling Solution是一种在单个细胞水平同时对转录组基因表达和适应性免疫受体库进行高通量测序的新技术,其基于10X Genomics平台,可以同时获取500-10000个单细胞的5’基因表达的数据和TCR/BCR的V(D)J全长序列,在肿瘤微环境、感染性疾病、器官移植后排斥、免疫治疗等研究领域中有着广泛的应用。在10X最新的产品中,单细胞免疫组库测序结合Feature Barcoding技术之后,还能同时检测和分析细胞表面标志物和抗原特异性等,从而加强免疫细胞表型分析,以及研究淋巴细胞和靶细胞之间的动态相互作用。
BD Rhapsody™ Single-Cell Analysis System的扩增和测序部分和10 x Chromium Single Cell Gene Expression Solution是一样的,两者的区别主要在于捕获单细胞的部分。
如图所示,这是BD捕获单细胞的方法。首先它使用的不是基于微流控芯片技术的双十字系统,而是蜂巢板。蜂巢板上每个孔的大小比一个细胞稍微大一些,可以同时放下一个细胞和一个凝胶微珠。它的操作是细胞悬液经注入孔注入后,自然沉降到反应孔中,随后, Beads 同样由注入孔注入,即可在单个反应孔中捕获其中的细胞。这里的 Beads 是刚性 Beads,捕获后在低温条件下可以保存3个月。Beads 上的序列结构与 10X Genomics 相似,可以捕获到游离 mRNA 的 polyA 尾。然后洗去多余的凝胶微珠,从而达到单细胞捕获的目的。
但是这样的操作会导致有的蜂巢孔是空的,而有的进去两个细胞,所以该系统装备了成像系统,在细胞捕获后,即可直观的看到细胞捕获数、细胞状态等信息。当这些统计的指标都是PASS的时候,就说明此次单细胞捕获是合格的,可以进行后续的操作了。当细胞捕获后,它的后续部分原理和10X就一样了。
数据处理:
标准预处理
1、QC和选择细胞进行进一步分析
2、规范化数据
3、识别高度可变的特征(特征选择)
4、缩放数据
主成分分析(PCA)
1、线性降维
2、确定数据集的“维度”
聚类细胞
数据呈现:
非线性降维(UMAP/tSNE)对低维度数据可视化处理,反映原始数据结构
寻找差异表达的特征(簇生物标志物),可视化比较某个基因在几个cluster之间的表达量
1. APPLICATION 单细胞测序的应用
单细胞转录组测序(scRNA-seq)
单细胞测序(scRNA-seq)对于制备的细胞悬液的细胞活性和细胞数目有着较高的要求,而且必须要求新鲜的样本,但是那些已经冻起来的样本库里面大量的珍贵样本,比如脑组织,心脏组织,肿瘤组织等都无法进行scRNA-seq,单细胞核转录组测序的出现极大的解决了这个问题。
单细胞核转录组测序(snRNA-seq)
单细胞核转录组测序(Single Nuclei RNA Sequencing,snRNA-seq):通过提取样本单细胞核,然后分离、标记细胞核,在单细胞水平研究核基因表达检测的技术。为脑组织、心脏、肾脏等复杂组织或一些珍稀冻存样品提供了单细胞水平研究应用平台,可挖掘更多潜在的致病细胞类型,更易于探讨肿瘤细胞异质性和致病机理。对免疫细胞类型不友好。
单细胞免疫组库测序
10X Genomics Single Cell Immune Profiling Solution是一种在单个细胞水平同时对转录组基因表达和适应性免疫受体库进行高通量测序的新技术,其基于10X Genomics平台,可以同时获取500-10000个单细胞的5’基因表达的数据和TCR/BCR的V(D)J全长序列,在肿瘤微环境、感染性疾病、器官移植后排斥、免疫治疗等研究领域中有着广泛的应用。在10X最新的产品中,单细胞免疫组库测序结合Feature Barcoding技术之后,还能同时检测和分析细胞表面标志物和抗原特异性等,从而加强免疫细胞表型分析,以及研究淋巴细胞和靶细胞之间的动态相互作用。